1.
Скачать данные Plasmodium falciparum chromosome 13: ftp://ftp.ncbi.nlm.nih.gov/genomes/Protozoa/Plasmodium_falciparum/NC_004317.gbk или скачиваем chtomosome 13 и ее аннотацию с ncbi. http://www.ncbi.nlm.nih.gov/genome/?term=Plasmodium+falciparum
Задание 2.
Прочесть позиции начал кодирующих регионов из gbk файла (CDS FEATURES). Позиции, в которых встречаются “<” или “>”, игнорировать(обозначают, что точные позиции начала или конца региона не известны).
Задание 3.
Вывести в файл последовательности нуклеотидов для начал сайтов трансляции(+- 10 нуклеотидов от первой позиции трансляции, т.е. всего 21 нуклеотид). Неизвестные нуклеотиды ‘N’ не надо учитывать. Если кодирующий регион идет “не последовательно”, то надо считывать только кодирующую область. (Например, если кодирующая область join(1000..1008,1200..1500), то начальная позиция +10 будет соответствовать координате 1201).
Задание 4.
Делим хромосому на 2 выборки (сайты трансляции, которые начинаются в первой половине хромосомы обучающий набор, остальные тестовый). Далее работаем с обучающей выборкой. Посчитайте частоту встречаемости нуклеотидов для каждой позиции сайта трансляции(PFM).
Отнесем к обучающему набору сайты трансляции, первая позиция находится принадлежит диапазону [1,1645935] (правая граница получена как 3291871/2 - половина длины нуклеотидной последовательности), остальные сайты трансляции отнесем к тестовому набору
| nucleotide \ position | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 | 13 | 14 | 15 | 16 | 17 | 18 | 19 | 20 | 21 |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
Sum |
382 |
382 |
382 |
382 |
382 |
382 |
382 |
382 |
382 |
382 |
382 |
382 |
382 |
382 |
382 |
382 |
382 |
382 |
382 |
382 |
382 |
A |
160 |
140 |
162 |
165 |
164 |
187 |
196 |
301 |
245 |
226 |
382 |
0 |
0 |
175 |
197 |
136 |
176 |
200 |
165 |
168 |
172 |
C |
27 |
18 |
22 |
28 |
20 |
22 |
52 |
17 |
25 |
20 |
0 |
0 |
0 |
26 |
59 |
33 |
31 |
34 |
45 |
49 |
50 |
G |
34 |
32 |
35 |
31 |
45 |
31 |
27 |
26 |
33 |
50 |
0 |
0 |
382 |
128 |
52 |
44 |
101 |
41 |
43 |
75 |
34 |
T |
161 |
192 |
163 |
158 |
153 |
142 |
107 |
38 |
79 |
86 |
0 |
382 |
0 |
53 |
74 |
169 |
74 |
107 |
129 |
90 |
126 |
| nucleotide \ position | 1.0 | 2.0 | 3.0 | 4.0 | 5.0 | 6.0 | 7.0 | 8.0 | 9.0 | 10.0 | 11.0 | 12.0 | 13.0 | 14.0 | 15.0 | 16.0 | 17.0 | 18.0 | 19.0 | 20.0 | 21.0 |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
A |
0.419 |
0.366 |
0.424 |
0.432 |
0.429 |
0.49 |
0.513 |
0.788 |
0.641 |
0.592 |
1.0 |
0.0 |
0.0 |
0.458 |
0.516 |
0.356 |
0.461 |
0.524 |
0.432 |
0.44 |
0.45 |
C |
0.071 |
0.047 |
0.058 |
0.073 |
0.052 |
0.058 |
0.136 |
0.045 |
0.065 |
0.052 |
0.0 |
0.0 |
0.0 |
0.068 |
0.154 |
0.086 |
0.081 |
0.089 |
0.118 |
0.128 |
0.131 |
G |
0.089 |
0.084 |
0.092 |
0.081 |
0.118 |
0.081 |
0.071 |
0.068 |
0.086 |
0.131 |
0.0 |
0.0 |
1.0 |
0.335 |
0.136 |
0.115 |
0.264 |
0.107 |
0.113 |
0.196 |
0.089 |
T |
0.421 |
0.503 |
0.427 |
0.414 |
0.401 |
0.372 |
0.28 |
0.099 |
0.207 |
0.225 |
0.0 |
1.0 |
0.0 |
0.139 |
0.194 |
0.442 |
0.194 |
0.28 |
0.338 |
0.236 |
0.33 |
Задание 5.
Используя найденные последовательности в пункте 2, постройте лого с помощью weblogo.
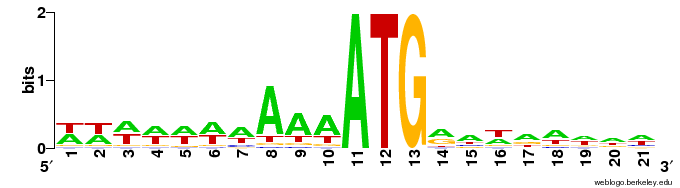
Задание 6.
Посчитать матрицу весов(position specific scoring matrix), используя результаты, полученные в пункте 4.
| Prior | Pos | 1.0 | 2.0 | 3.0 | 4.0 | 5.0 | 6.0 | 7.0 | 8.0 | 9.0 | 10.0 | 11.0 | 12.0 | 13.0 | 14.0 | 15.0 | 16.0 | 17.0 | 18.0 | 19.0 | 20.0 | 21.0 |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
1.0 |
Sum |
-0.108 |
-0.265 |
-0.165 |
-0.129 |
-0.093 |
-0.215 |
0.046 |
-0.526 |
-0.215 |
-0.1 |
-1.524 |
-1.528 |
-0.301 |
0.362 |
0.328 |
0.065 |
0.359 |
0.028 |
0.178 |
0.431 |
0.128 |
0.407 |
A |
0.015 |
-0.05 |
0.022 |
0.031 |
0.028 |
0.097 |
0.123 |
0.385 |
0.254 |
0.205 |
0.548 |
-0.692 |
-0.692 |
0.062 |
0.126 |
-0.064 |
0.065 |
0.135 |
0.031 |
0.04 |
0.053 |
0.092 |
C |
-0.118 |
-0.274 |
-0.202 |
-0.102 |
-0.237 |
-0.202 |
0.22 |
-0.293 |
-0.151 |
-0.237 |
-0.688 |
-0.688 |
-0.688 |
-0.134 |
0.297 |
-0.026 |
-0.056 |
-0.011 |
0.136 |
0.185 |
0.197 |
0.093 |
G |
-0.02 |
-0.049 |
-0.006 |
-0.064 |
0.127 |
-0.064 |
-0.126 |
-0.142 |
-0.035 |
0.187 |
-0.692 |
-0.692 |
1.773 |
0.835 |
0.21 |
0.114 |
0.655 |
0.076 |
0.102 |
0.443 |
-0.02 |
0.409 |
T |
0.015 |
0.108 |
0.022 |
0.006 |
-0.01 |
-0.047 |
-0.171 |
-0.475 |
-0.284 |
-0.254 |
-0.693 |
0.544 |
-0.693 |
-0.401 |
-0.305 |
0.04 |
-0.305 |
-0.171 |
-0.091 |
-0.238 |
-0.102 |
Задание 7.
Используя матрицу весов, постройте гистограмму scores для
-
известных начал CDS в обучающем наборе (из пунктов 12)
-
всех подпоследовательностей во всей хромосоме. Приблизить это распределение нормальным.
-
сводный график (зеленым - гистограмма a, красным - гистограмма b)
-
сводный график 2 (зеленым - гистограмма a, красным - гистограмма b - отличие в том, что данные для гистограммы b берутся по последовательности, а не генерируются)
Задание 8.
По данным, полученным в предыдущем пункте, постройте график FP, FN в зависимости от отсечки для score, по которой определяется, что последовательность в хромосоме это CDS.
То же самое нормализованное:
Задание 9.
С использованием значения отсечки, которое кажется вам разумным, проведите поиск CDS во второй половине хромосомы и проанализируйте результат (FP, FN, sensitivity, specificity, and PPV).
Я взяла отсечку 2.5, и вот что получилось: